qPCR
This video tests your knowledge on how to execute crucial steps of RNA isolation, cDNA synthesis and qPCR.
- Tineke Bijl
-
(0)
- 0 enrolled students
Description
Non-interactive video download link (mp4).
The script that has been used to create the video shown above can be downloaded as excel script file.
| 1 | Als 2 samenwerkende teams gaan jullie de 96-wells qPCR plaat vullen volgens deze sample indeling die door de assistenten wordt uitgedeeld. Mocht er per ongeluk een sample in een andere well gepipetteerd worden, schrijf dit dan goed op én geef het door aan de assistenten. De ruwe data van de qPCR worden nl. op Blackboard gezet zodat andere teams deze ook kunnen gebruiken als vergelijkingsmateriaal. |
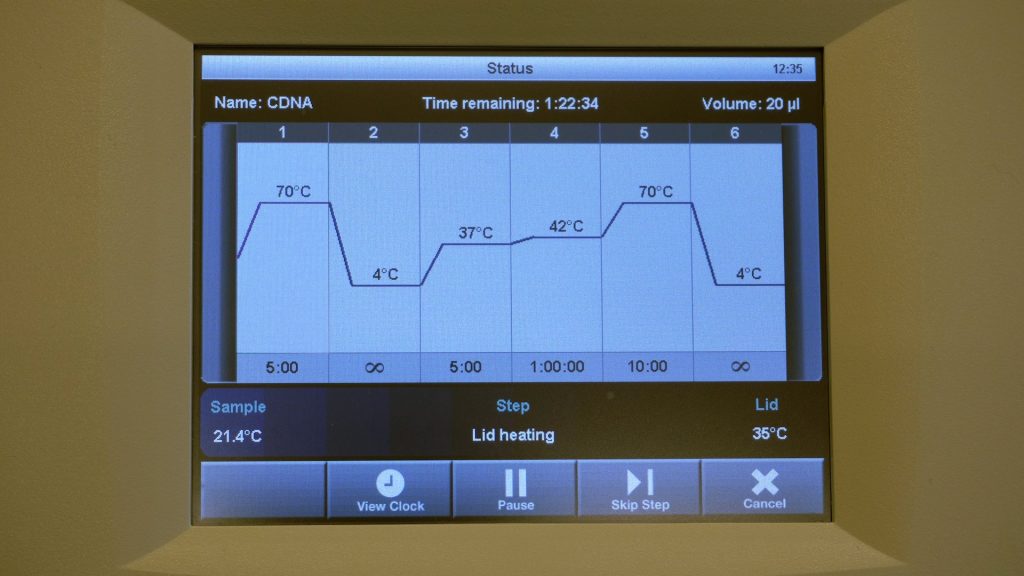
| 2 | Maak per gen een qPCR mastermix met daarin per sample:
Maak genoeg voor 10 samples per mastermix voor elk gen. Voor GAPDH maak je genoeg voor 12 samples, aangezien de no-RT hier ook bij hoort. NB: Houd deze volgorde bij het pipetteren aan. Meng de qPCR mastermix door een paar keer tegen het epje te tikken (niet centrifugeren) en bewaar op ijs en in het donker zolang je er niet mee pipetteert (SensiMix is lichtgevoelig). |
| 3 | Er mag alleen op de zijkant van de plaat geschreven worden! Pipetteer, volgens de uitgedeelde sample-indeling, 4 µL cDNA per well in de PCR-plaat. |
| 4 | Pipetteer, volgens de uitgedeelde sampleindeling, 6 µL van de juiste qPCR mastermix in de wells van de PCR-plaat. |
| 5 | Als alle koppels de plaat hebben gevuld: laat de gehele plaat goed afsluiten met plakfolie. De assistenten starten het qPCR-apparaat zodra dat vrij is:
NB Lever je cDNA weer in bij de assistent. |
| 6 | Na afloop wordt de qPCR data op Brightspace gezet in de map ‘resultaten qPCR’. Bedenk hoe je de output (Ct = threshold Cycle) kunt omrekenen naar de relatieve genexpressie. Je kunt deze tabel gebruiken voor het verwerken van de Ct waardes. |
The sample that is placed in the QPCR machine contains DNA. Which of the following quote(s) is/are true?
Feedback if incorrect: Why do we use different primers? Feedback if correct: Correct! In the previous step, the mRNA is reverse transcripted to cDNA. We are interested in specific genes, the rest of the DNA is not important for our experiment. That is the reason why these quotes are correct. With the primers in the reaction mixture, specific genes can be selected to be amplified in the RT-PCR. |
What range of Ct levels are considered illustrative for abundant, moderate and minimal amounts of target nucleic acid respectively? Think about how the amplification step works. What does a high number of cycles actually mean? And what does a low number of cycles mean? What does this say about the starting material (amount)?
|
Every time there is twice as much DNA, your Ct values decrease by 1 and will not halve. This indicates the calculations are in logarithm base 2. Why do we use a Log2 when normalizing the expression of genes?
Feedback if correct: Correct! The correct answers indicate two major points; the fold change concerning cDNA amplification does not follow a normal distribution and while plotting the data in Log2 scale both up- and downregulation will look as equally significant. While the mathematics behind the use of Log2 are more complex, it is important to understand that plotting the data with a Log2 transformation is more appropriate for the qPCR data analysis. |
Na het analyseren van de qPCR data blijkt dat na behandeling van bortskankercellen met stof X de relatieve genexpressie (oftewel fold change) van gen y 5.0 is. Stelling: Stof X remt de expressie van gen y; deze is 5 maal zo laag als in onbehandelde cellen.
|
Na behandeling van bortskankercellen met stof Z is de relatieve genexpressie (oftewel fold change) van gen y 0.1 is. Wat zegt deze waarde?
Feedback if b: Juist! De relatieve genexpressie van onbehandelde cellen is 1 (ΔΔCt = 0, 2^-ΔΔCt = 1) en een relatieve genexpressie/fold change van 0.1 wil zeggen dat de expressie 0.1 keer zo hoog is (oftewel 1/0.1 = 10 keer zo laag is) als in onbehandelde cellen. Feedback if d: Ook juist maar niet zo mooi geformuleerd als antwoord B! De relatieve genexpressie van onbehandelde cellen is 1 (ΔΔCt = 0, 2^-ΔΔCt = 1) en een relatieve genexpressie/fold change van 0.1 wil zeggen dat de expressie 0.1 keer zo hoog is (oftewel 1/0.1 = 10 keer zo laag is) als in onbehandelde cellen. Feedback if a or c: Bedenk goed waarmee je de genexpressie van behandelde cellen vergelijkt. Wat is de relatieve genexpressie/fold change van onbehandelde cellen? En wat gebeurt er met de relatieve genexpressie/fold change/ΔΔCt als de genexpressie toe- of afneemt (vul evt. voorbeeld Ct waarden in in de formules). Feedback if b and d: Antwoorden zijn beide juist omdat ze in feite hetzelfde zijn. Antwoord B is echter beter geformuleerd. De relatieve genexpressie van onbehandelde cellen is 1 (ΔΔCt = 0, 2^-ΔΔCt = 1) en een relatieve genexpressie/fold change van 0.1 wil zeggen dat de expressie 0.1 keer zo hoog is (oftewel 1/0.1 = 10 keer zo laag is) als in onbehandelde cellen. |
Je voert een real-time PCR uit met verschillende samples. Bij het analyseren van de data valt op dat één sample een aanmerkelijk lagere Ct waarde heeft dan de andere samples. Is de volgende stelling met betrekking tot dit sample juist of onjuist? Stelling 1: Het fluorescentiesignaal van dit sample bereikt eerder de drempelwaarde dan het fluorescentiesignaal van de andere samples. Juist/onjuist Stelling 2: Het sample bevat aanmerkelijk meer cDNA in vergelijking met de andere samples. Juist/onjuist
Feedback if correct: De Ct waarde geeft aan wanneer het fluorescentiesignaal de drempelwaarde bereikt. Een lagere Ct waarde geeft dus aan dat de drempelwaarde eerder is bereikt dan bij een hogere Ct waarde. De fluorescentie wordt sterker naar mate er meer qPCR product in het sample aanwezig is. Wanneer de drempelwaarde sneller wordt bereikt, betekent dit dat er meer qPCR product aanwezig is in het sample waardoor de fluorescentie hoger is dan bij de andere samples en de Ct waarde lager is. Feedback if incorrect: Ga nog eens na wat een drempelwaarde aangeeft. |
Het epje wat in de PCR machine geplaatst wordt, bevat DNA. Welke stelling(en) over dit DNA zijn/is juist?
Feedback if correct: Juist! Stelling 2 is niet juist want alleen de genen van interesse worden geamplificeerd. Met behulp van de primers in de reactiemix selecteren we de genen die vervolgens geamplificeerd worden. Feedback if incorrect: Bedenk wat er precies gebeurt tijdens de qPCR. Wat is je uitgangsmateriaal en wat meet je? |
Bij het koken van een ei worden de eiwitten gedenatureerd, waarbij ze hun oorspronkelijke functie en structuur verliezen. Bij de amplificatie van DNA wordt het DNA in het sample gedenatureerd, wat gebeurt door het sample met DNA te verwarmen tot 94⁰C (smeltpunt van DNA). Welke stelling(en) over de denaturatie van DNA is/zijn juist?
Correct = yes
Correct = yes Feedback if correct: Juist! Bij denaturatie van het DNA verliest het DNA de oorspronkelijke structuur en laten de twee strengen los van elkaar. De denaturatie van DNA is nodig om een primer te laten binden en het DNA te kunnen amplificeren tijdens de (q)PCR. Als de denaturatie van DNA niet reversibel zou zijn, is het niet mogelijk om enkelstrengs DNA te kopiëren, aangezien het bij kopiëren weer dubbelstrengs DNA wordt. In het lichaam treedt het loslaten van de twee strengen DNA ook op, denk maar aan de replicatievork bij de DNA transcriptie. Na de transcriptie ‘sluit’ het DNA, waarbij de twee strengen weer bij elkaar komen. De denaturatie van DNA is dus reversibel.
Feedback if not a: Binden primers op enkelstrengs – of dubbelstrengs DNA? Feedback if not b: Denk eens aan de replicatievork bij de DNA-transcriptie. Wat gebeurt er bij het kopiëren van DNA?
|
Na het toevoegen van primers aan het gedenatureerde DNA binden de primers aan het enkelstrengs DNA om de duplicatie van het desbetreffende DNA te starten. Je zou verwachten dat deze binding niet mogelijk is, aangezien de twee strengen van het DNA loslaten van elkaar. Echter, voordat de primers kunnen binden moet de temperatuur verlaagd worden naar 50-65⁰C. Waarop wordt deze temperatuur gebaseerd?
Feedback if correct: Juist! De lengte en nucleotide samenstelling van een primer bepalen de bindingskracht aan DNA en daarmee de specifieke temperatuur om optimaal aan DNA te binden. Bij een te lage temperatuur binden primers ook aan DNA als er met de tegenoverliggende nucleotiden een of meerdere mismatches zijn; dan kunnen ook andere (= niet-specifieke) genen geamplificeerd worden. De bindingskracht van de primer en de temperatuur van 64⁰C worden zo gekozen dat de primer dan nog nét aan DNA kan binden. Dit kan alleen als álle nucleotiden gebonden zijn met het juiste nucleotide tegenover zich; al met één mismatch is de bindingskracht te laag. Hierdoor wordt de maximale specificiteit voor het gen van interesse bereikt. Feedback if b and c: Denk eens aan de factoren die bepalen of een primer specifiek aan DNA kan binden. Feedback if a and c: Wat gebeurt er met de sterkte van de binding tussen nucleotiden bij een temperatuur lager dan 50⁰C? Feedback if a and b: Wat gebeurt er met de sterkte van de binding tussen nucleotiden bij een temperatuur hoger dan 65⁰C? Feedback if incorrect: Ga nog eens na waarop deze temperatuur gebaseerd is. |
Download zip to import in LabBuddy.
Please make sure that your product exists and valid for this course
- Skill levelIntroduction video
- CategoryMolecular biology
Related videos
-
Free
qPCR
-
Free
cDNA synthesis
-
Free
RNA isolation
Copyright information
This video is created by Leiden Academic Centre for Drug Research (LACDR), Faculty of Science at Leiden University under a open Creative Commons Attribution-NonCommercial-ShareAlike 4.0 International License. When using this video in its original version please refer to www.labprep.video. When adapting the video, mention the source ‘adapted based on the original version that is created by the labprep.video team’. It is not allowed to use the video for commercial purposes without consultation with the creators. You can contact us via info@labprep.video.